1200+
Cribles complétés
60+
Collaborations
ChemoGenix
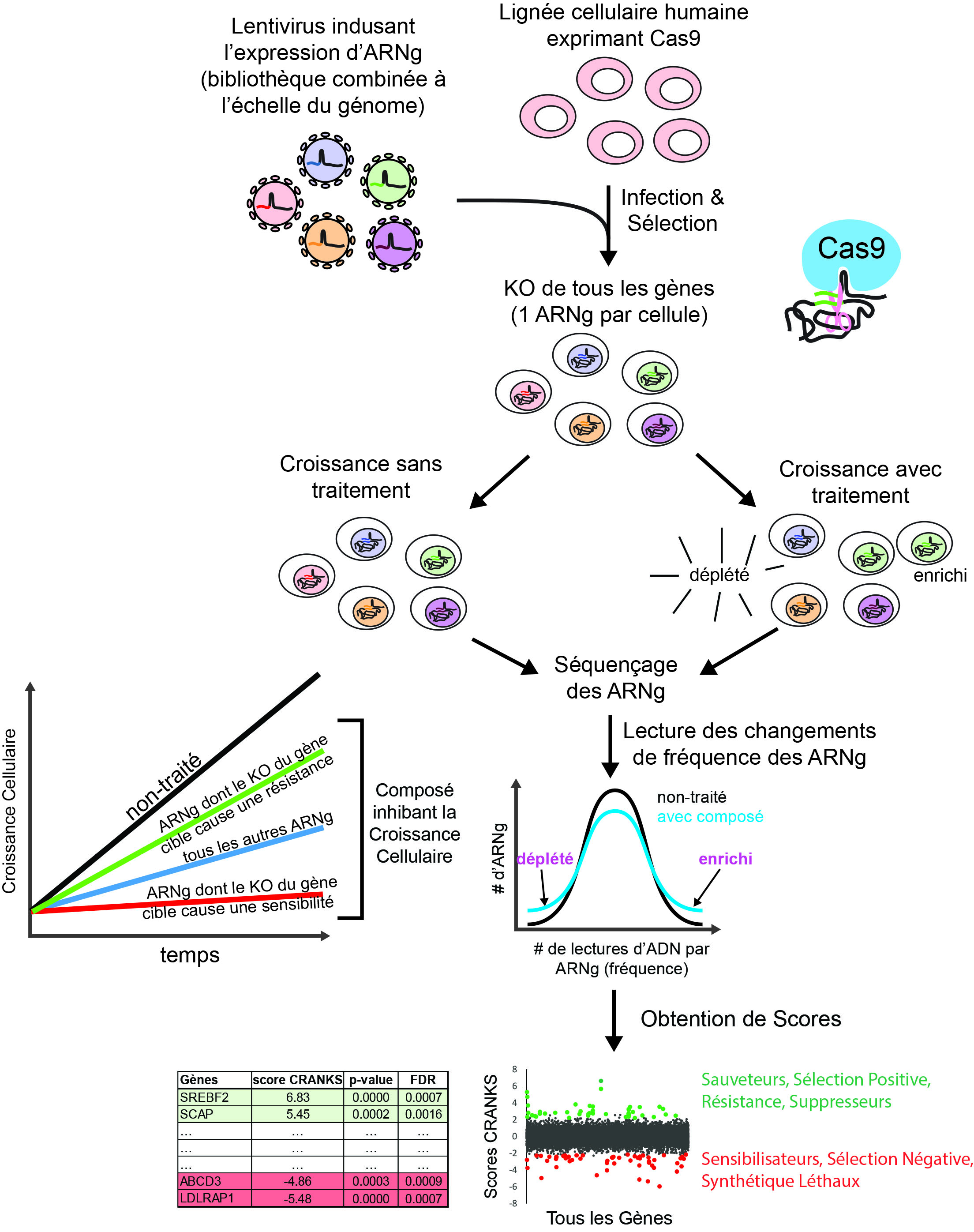
ChemoGenix est la plateforme de criblage CRISPR/Cas9 combinée (pooled) chimiogénomique de l’IRIC, Université de Montréal.
Le 17 mai 2024, l’Université de Montréal (UdeM) a signé une entente non-exclusive avec ERS Genomics (ERS) lui procurant accès à son porte-folio de brevets
CRISPR/Cas9. Avec l’acquisition de cette licence, l’UdeM lance maintenant la plateforme ChemoGenix utilisant la technologie CRISPR/Cas9.
Nous nous spécialisons dans l’identification de tous les gènes causant une vulnérabilité ou une résistance génétique en réponse à des composés bioactifs.
Ces signatures génétiques peuvent alors servir à établir le mécanisme d’action (MOA) du composé et trouver de nouvelles fonctions de gènes.
Contactez-nous pour apprendre comment nous pourrions collaborer sur vos projets.